当前位置 > 海洋三所 > 科学研究 > 科研成果 > > 正文内容
自然资源部第三海洋研究所海洋生物多样性研究室邢炳鹏副研究员在我国软体动物系统演化与生态适应机制方面取得新进展。相关成果以 “Comparative Genomic and Mitochondrial Phylogenetic Relationships of Ovulidae (Mollusca: Gastropoda) Along the Chinese Coast” 为题,发表于 Ecology and Evolution(JCR Q1 区,中科院大类 2 区)。
●研究背景
梭螺科(Ovulidae)与宝贝科(Cypraeidae)亲缘关系密切,主要分布于热带—亚热带浅海生态系统,并长期与软珊瑚、皮革珊瑚及黑珊瑚等宿主形成高度专一的寄生关系。这种强烈的宿主依赖性被认为是驱动梭螺科形态多样化和快速分化的重要生态因素,但宿主选择如何在分子层面塑造其演化轨迹仍缺乏系统认识。现有研究多停留在形态描述或单基因证据层面,难以有效区分宿主诱导的表型可塑性与真实的系统分化,从而限制了对其演化与生态适应机制的深入理解。本研究基于线粒体基因组的系统发育重建及选择压力分析,为我国近海梭螺类群多样性格局、演化动力及其生态适应机制提供了新的研究视角。
●研究结果
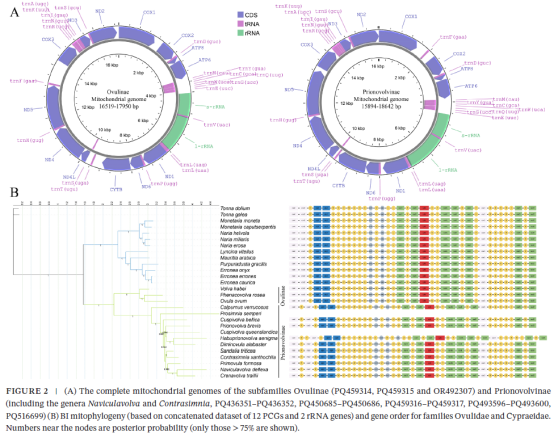
本研究首次记录 Habuprionovolva aenigma 在我国海域的分布。基于线粒体基因组构建的系统发育结果显示,tRNA 基因排列为梭螺科亚科分化与分类修订提供了关键分子证据。研究支持将 Naviculavolva 属与 Contrasimnia 属调整归入 Prionovolvinae科,并指出 Cuspivolva 属不具单系性,其中 Cuspivolva queenslandica 更接近 Primovula属,提示该属亟需系统修订。系统发育分析进一步揭示,Naviculavolva 属与 Crenavolva 属为稳定的姐妹群,其关系联合 Contrasimnia属的系统位置,有力反驳了既有 Simniinae 单系性的观点;同时,Phenacovolva 属与 Ovula 属的姐妹关系亦纠正了传统分类假说。

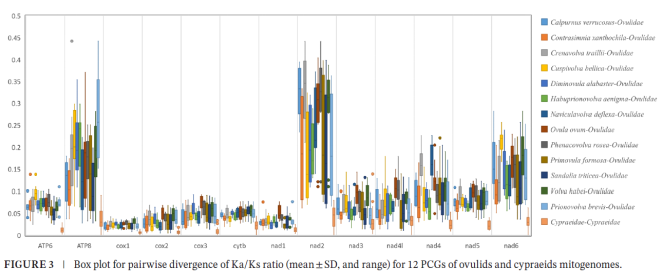
研究首次发现,除 Ovula属、Volva属 与 Phenacovolva 属外,其余梭螺类群均具有独特的tRNA 排列模式,指示线粒体基因重排可能发生于 Prionovolvinae 与 Ovulinae 分化之后。选择压力分析显示,宝贝科线粒体蛋白编码基因承受的选择压力显著高于梭螺科,提示高度宿主专化的生态策略可能降低了梭螺科的进化竞争压力。
选择压力分析显示,梭螺科线粒体蛋白编码基因的 Ka/Ks 分布特征与宝贝科存在显著差异,整体上宝贝科承受的选择压力明显高于梭螺科。研究推测,这一差异可能与梭螺科高度特化的宿主取食策略有关:稳定而专一的宿主资源可能降低了生态位竞争强度,从而减弱了线粒体基因层面的选择压力。这一结果从分子进化角度揭示了宿主专化、生态策略与基因组演化之间的潜在耦合机制。
该论文第一作者为北京师范大学—海洋三所联合培养博士研究生吴穹,通讯作者为邢炳鹏副研究员,共同作者包括项鹏研究员等。该项研究工作得到了国家重点研发计划项目 (2022-24) 、福建省自然科学基金(2023J01208)、厦门市自然科学基金(3502Z20227252)的资助。