当前位置 > 海洋三所 > 科学研究 > 科研成果 > > 正文内容
近日,我所海西站郑新庆研究员领衔的珊瑚研究团队联合上海大学杨明研究员团队,在海洋三所蔡灵高级工程师、周曦杰助理研究员和上海大学—海洋三所联合培养博士研究生张琦的共同参与下,成功完成全球首个卷曲黑星珊瑚(Oulastrea crispata)的高质量全基因组测序、组装与注释分析,获得该物种染色体级参考基因组。
尽管此前已有针对鹿角珊瑚属等其他造礁石珊瑚的基因组测序成果,然而,由于造礁石珊瑚组织中共生藻和杂质较多、长片段高质量DNA提取困难等原因,造礁石珊瑚的的基因组学研究长期受限。建立造礁石珊瑚高质量参考基因组对于解析珊瑚的适应进化、共生关系以及环境胁迫响应机制具有重要意义。
卷曲黑星珊瑚是一种典型的边缘型造礁石珊瑚,在低温、低光、高浊度等极端环境中具有独特的生存优势。此前团队沿华南沿岸至宁波渔山列岛均发现其分布,表明其具有极宽的生态幅,是研究珊瑚在极端或高纬环境中生存策略的重要模式物种。卷曲黑星珊瑚该高质量基因组的获得,不仅为深入探索边缘珊瑚共生体的环境适应机制提供了关键数据资源,也为全球珊瑚礁保护与生态修复提供了新的分子依据。
课题组通过创新多重胁迫联合技术,成功获得活体白化卷曲黑星珊瑚(不含虫黄藻基因组)的纯珊瑚组织样品。利用PacBio HiFi三代测序、Illumina二代测序和Hi-C空间组装等多组学策略,成功构建卷曲黑星珊瑚高质量基因组。基因组分析结果显示,该物种基因组大小约417.08 Mb,杂合度3.92%,重复序列占比49.02%,为“低重复、高杂合”型基因组。组装后基因组总长度达622.35 Mb,contig N50为18.06 Mb,BUSCO完整性达96.2%,显示组装质量优异。通过Hi-C辅助组装,共将494.70 Mb序列(占比90.69%)锚定至14条染色体上,最大染色体长度为55.71 Mb,标志着该基因组达到国际高标准的染色体级组装水平。
下一步,研究团队将基于该基因组开展功能基因注释、比较基因组分析及调控网络研究,揭示卷曲黑星珊瑚的适应性进化机制,为珊瑚礁生态系统保护和修复提供科学支撑。

图1 卷曲黑星珊瑚正常(a)和白化活体(b和c)样品
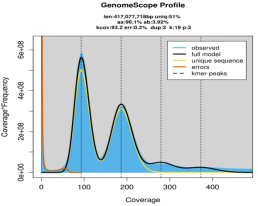
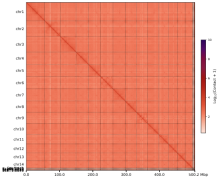
图2 卷曲黑星珊瑚基因组survey的K-mer 深度与频率分布图(Kmer=19)以及染色体区间互作关系图
该项研究工作得到了自然资源部海峡西岸海岸带生态系统野外科学观测研究站开放共享航次的支持,以及国家重点研发计划(2022YFF0802204)、自然资源部第三海洋研究所基本科研业务费(2019017)、国家自然科学基金(42376110,U24A20607)、福建省自然科学基金杰青项目(2023J06043)基金项目的资助。